Aujourd’hui, dans tous les domaines de la biologie le nombre de données ne cesse d’augmenter et les chercheurs ont besoin d’outils pour pouvoir les traiter. Deux langages vont principalement être utilisés, Python et R. Python est plus souvent utilisé en génétique et phylogénie tandis qu’on privilégie R dans les domaines de l’écologie et de la biologie comparative.
R, c’est d’abord le nom d’un langage de programmation, développé à partir du langage S de John Chambers dans le cadre du projet GNU (dont est notamment issu le système d’exploitation Linux). Codé initialement par Robert Gentleman et Ross Ihaka en 1993, une équipe de 20 personnes maintient le projet. R est aussi un logiciel codé évidemment dans le langage R, mais aussi en C, C++ et en Fortran (la connaissance de ces autres langages peut être utile en cas d’opérations qui demandent beaucoup de pouvoir de calcul).
Le premier avantage évident de R est d’être un logiciel gratuit en OpenSource, ce qui est non négligeable en vue des contraintes budgétaires associées à la recherche.
Un autre avantage est la grande quantité de paquets disponibles pour adapter le logiciel à vos besoins. En 2015, on compte par exemple plus de 900 paquets dédiés à R sur Bioconductor (site de ressources pour la bioinformatique). Avec R vous avez aussi la possibilité d’effectuer une grande quantité de tests statistiques, (Student, F, Z, Χ² et bien d’autres encore).

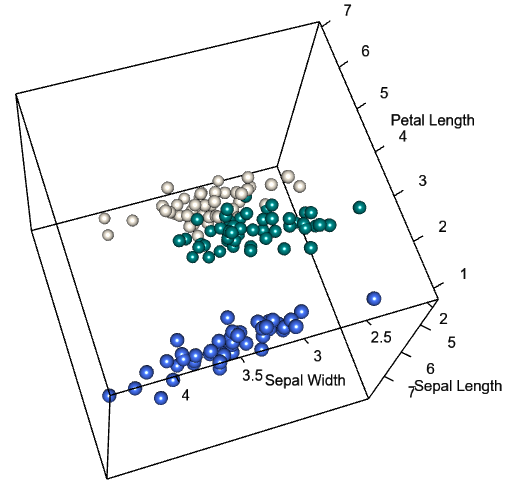
Un dernier atout de R, c’est sa capacité à réaliser une grande gamme de graphiques. Il est même possible de réaliser des graphiques en 3D, cependant certains déconseillent leur utilisation à part dans des contextes particuliers de par leur manque de lisibilité. Ainsi, vous avez à gauche un nuage de points (modélisé avec le paquet rgl), basé sur le jeu de données Iris, qui est déjà présent sur le logiciel. Il est difficile d’estimer ici où se placent les points, cependant le fait de le rendre interactif (avec la possibilité de le tourner) peut pallier ce problème.
Un paquet pour utiliser les données de la phylogénie et de l’évolution a été développé pour la première fois en 2003 par des chercheurs de Montpellier et de Munich, le paquet APE (Analysis of Phylogenetics and Evolution). Il permet notamment de dessiner des arbres phylogénétiques plus ou moins complexes comme on peut le voir ci-dessous.
Le paquet permet aussi d’estimer des phylogénies selon plusieurs méthodes (NJ, UPGMA, Parcimonie, etc…)
L’interface de base de R n’est cependant pas la plus accueillante. C’est pourquoi est développé en 2009 le logiciel RStudio. RStudio permet d’avoir une interface utilisateur plus facile d’accès. A savoir que RStudio est aussi un logiciel gratuit en OpenSource. De plus, la compagnie qui l’opère propose d’autres services comme du support technique ainsi que la possibilité d’accéder à des serveurs dématérialisés (utiles par exemple dans un cadre d’apprentissage).
Ci-dessus se trouve un script simple que j’ai tapé pour cet article.
Comme vous pouvez le voir, l’interface est divisée en 4 fenêtres. Il faut d’abord taper le code souhaité dans la fenêtre script (1). Puis quand on exécute le script, on voit le résultat dans la fenêtre de console (2). Les objets que l’on crée sont visibles dans l’environnement (3) tandis que les graphiques s’affichent dans la fenêtre qui leur est dédiée (4).
R reste un logiciel qui peut prendre un certain temps à maîtriser, il nécessite aussi une connaissance des outils statistiques impliqués, cependant de par sa popularité il y a grand nombre de ressources disponibles pour vous aider dans cet apprentissage. De plus, un certain nombre de masters ont maintenant des formations dédiées à R dans leur parcours.
Matthias Rudeanu
Sources :
- Paradis, Emmanuel, Julien Claude, and Korbinian Strimmer. “APE: Analyses of Phylogenetics and Evolution in R Language.” Bioinformatics 20.2 (2004): 289–290. Web.
- Paradis, Emmanuel. Analysis of Phylogenetics and Evolution with R. 2nd ed. New York, NY: Springer New York, 2012. Web.
- Cohen, Yosef, and Jeremiah Y. Cohen. Statistics and Data with R an Applied Approach through Examples . Chichester, U.K: Wiley, 2008. Print.
- Lafaye de Micheaux Pierre. Le logiciel R : maîtriser le langage, effectuer des analyses (bio)statistiques. 2e édition. Cachan: Lavoisier-Hermes, 2017. Print.
- Wright AM, Schwartz RS, Oaks JR et al. The why, when, and how of computing in biology classrooms [version 2; peer review: 2 approved]. F1000Research 2020, 8:1854 (https://doi.org/10.12688/f1000research.20873.2)
- 3D | the R Graph Gallery [WWW Document], n.d. URL https://www.r-graph-gallery.com/3d (consulté le 18/12/21).
- About RStudio [WWW Document], n.d. URL https://rstudio.comhttps://www.rstudio.com/about/ (consulté le 17/12/21).
- ape – home page [WWW Document], n.d. URL http://ape-package.ird.fr/index.html (consulté le 17/12/21).
- Bioconductor [WWW Document], n.d. URL https://www.bioconductor.org (consulté le 17/12/21).
- Guillaume, 2020. Logiciel R : un outil de Data Science pour les naturalistes. Louernos Nature. URL https://louernos-nature.fr/logiciel-r-ecologie-naturaliste/ (consulté le 14/12/21).
- R Programming – Manuals [WWW Document], n.d. URL http://manuals.bioinformatics.ucr.edu/home/programming-in-r (consulté le 14/12/21).
- R: What is R? [WWW Document], n.d. URL https://www.r-project.org/about.html (consulté le 14/12/21).
- University of Michigan Computational Medicine and Bioinformatics, 2020. An introduction to the R programming language for Bioinformatics students – Part 1/2 [WWW Document]. URL https://www.youtube.com/watch?v=bekFrlW0gww (consulté le 17/12/21).
Sources photographiques :
- Figure 1 : 3D | the R Graph Gallery [WWW Document], n.d. URL https://www.r-graph-gallery.com/3d (consulté le 18/12/21).
- Figure 2 : Paradis, Emmanuel. Analysis of Phylogenetics and Evolution with R. 2nd ed. New York, NY: Springer New York, 2012. Web.

Comments are closed